Sztuczna inteligencja DeepMind rozkodowała strukturę 200 milionów białek
Nauka zna około 200 milionów białek, ale do tej pory naukowcy rozszyfrowali budowę tylko ułamka z nich. Dzięki sztucznej inteligencji DeepMind i programowi AlphaFold w dwa lata udało się poznać struktury całej reszty. To otwiera drogę do opracowania nowych leków i technologii medycznych.
Białka to główny budulec życia, a ich zbudowane z aminokwasów struktury są niesamowicie złożone. Gdy poznamy ich budowę, można pokusić się o zrozumienie mechanizmu działania oraz tego, jak można go modyfikować. Teoretycznie można sekwencjonować DNA, ale przewidywanie jak jego łańcuchy oddziałują na siebie tworząc strukturę 3D, jest bardzo kłopotliwe. Dlatego właśnie naukowcy do 2020 roku radzili sobie bardzo słabo z rozkodowywaniem ich budwy.
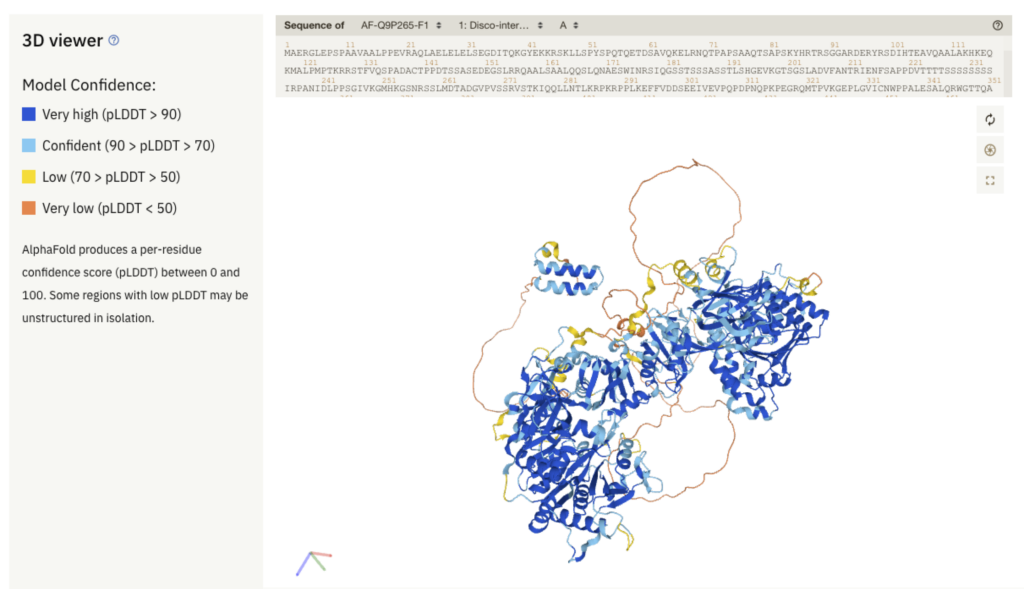
Wszystko zmieniło się wraz z końcem 2020 roku, gdy grupa naukowców pracująca przy projekcie sztucznej inteligencji DeepMind ogłosiła opracowanie programu AlphaFold, który pozwolił na błyskawiczne opracowanie olbrzymich ilości danych wymaganych przy określaniu struktury białek. Od tego czasu algorytm przebrnął przez kody DNA każdego organizmu, którego genom poznaliśmy i określił struktury białek stanowiących ich budulec.
W zeszłym roku zespół DeepMind opublikował w otwartej bazie danych struktury białek dla 20 gatunków – w tym prawie wszystkich 20 000 białek wyrażanych przez człowieka. Teraz naukowcy zakończyli pracę i opublikowali bazę struktur dla ponad 200 milionów białek.
DeepMind pomaga w tworzeniu nowych szczepionek
Zasadniczo można myśleć o tym, że obejmuje ona cały wszechświat białek. Zawiera przewidywane struktury dla roślin, bakterii, zwierząt i wielu innych organizmów, otwierając ogromne możliwości dla AlphaFold, aby miał wpływ na ważne kwestie, takie jak zrównoważony rozwój, bezpieczeństwo żywnościowe i choroby, którym nie poświęcaliśmy należytej uwagi. – powiedział Demis Hassabis, założyciel i szef DeepMind.

Naukowcy już wykorzystują niektóre z wcześniejszych przewidywań, aby pomóc w opracowaniu nowych leków. W maju badacze pod kierownictwem prof. Matthew Higginsa z Uniwersytetu w Oksfordzie ogłosili, że wykorzystali modele AlphaFold do określenia struktury kluczowego białka pasożyta malarii i opracowania miejsca, w którym prawdopodobnie będą wiązać się przeciwciała mogące zablokować jego transmisję.
Wcześniej używaliśmy techniki zwanej krystalografią białek, aby dowiedzieć się, jak wygląda ta cząsteczka, ale ponieważ jest ona dość dynamiczna i porusza się, nie mogliśmy się z nią uporać. Gdy wzięliśmy modele AlphaFold i połączyliśmy je z dowodami eksperymentalnymi, nagle wszystko nabrało sensu. To spostrzeżenie zostanie teraz wykorzystane do zaprojektowania ulepszonych szczepionek, które spowodują wytworzenie najsilniejszych przeciwciał blokujących transmisję – powiedział Higgins.
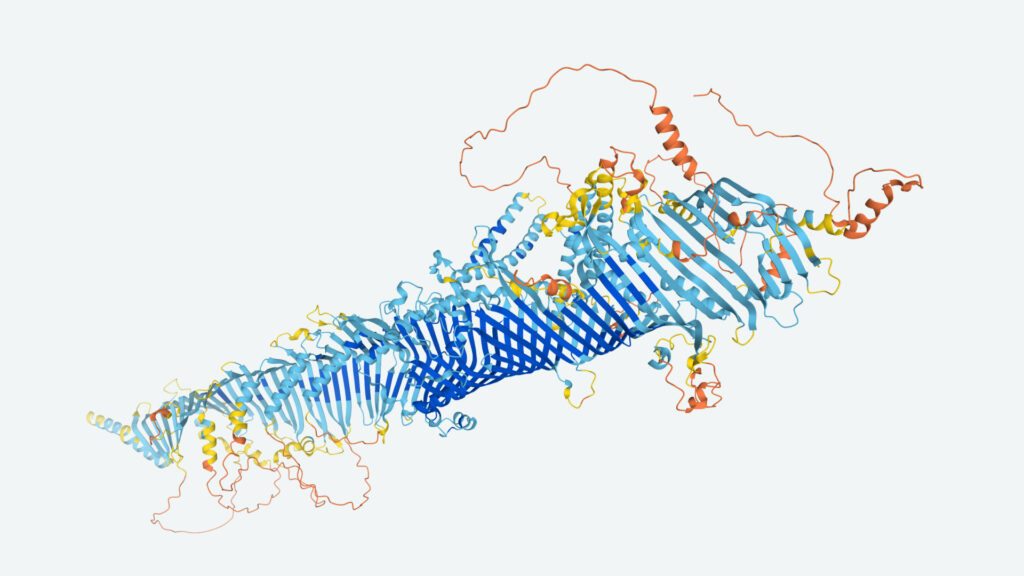
Modele AlphaFold są również wykorzystywane przez naukowców z University of Portsmouth’s Centre for Enzyme Innovation, w celu zidentyfikowania enzymów ze świata przyrody, które mogłyby być dostosowane do rzkładania i recyklingu tworzyw sztucznych. „Zajęło nam dość dużo czasu, aby przejść przez tę ogromną bazę danych struktur, ale otworzył ten cały wachlarz nowych trójwymiarowych kształtów, których nigdy wcześniej nie widzieliśmy, a mogłyby one rzeczywiście rozłożyć tworzywa sztuczne. Następuje całkowita zmiana paradygmatu. Możemy naprawdę przyspieszyć, – a to pomaga nam skierować cenne zasoby na rzeczy, które mają znaczenie – powiedział prof John McGeehan, który kieruje pracą.
Prof. Dame Janet Thornton, lider grupy i starszy naukowiec w Europejskim Instytucie Bioinformatyki Laboratorium Biologii Molekularnej, powiedział – Przewidywania struktury białek AlphaFold są już wykorzystywane na niezliczoną ilość sposobów. Spodziewam się, że ta najnowsza aktualizacja wywoła lawinę nowych i ekscytujących odkryć w nadchodzących miesiącach i latach, a to wszystko dzięki temu, że dane są otwarte dla wszystkich.
Źródło: Guardian

Miłośnik nowoczesnych technologii, głównie nowych rozwiązań IT. Redaktor w czasopismach Gambler, Enter, PC Kurier, Telecom Forum, Secret Service, Click!, Komputer Świat Gry, Play, GameRanking. Wiele lat spędził w branży tłumaczeniowej – głównie gier i programów użytkowych. W wolnych chwilach lata szybowcem, jeździ na rowerze i pochłania duże ilości książek.